实验二十五 启动子序列的克隆——染色体步移法
由于拟南芥基因组序列已测序完成,研究其某个基因启动子时,可以根据已知的基因序列设计引物,直接克隆基因的启动子。但是对于烟草、油菜等植物材料,由于没有已知的基因组序列,在克隆某个基因启动子时需要利用染色体步移(genome walking)。
【实验目的】
1.了解染色体步移的基本原理。
2.学习基于热不对称PCR的染色体步移技术及步骤。
【实验原理】
染色体步移的方法中常用的是环状PCR,包括反向PCR、锅柄PCR、连接介导PCR、热不对称PCR(Tail-PCR)、单侧寡聚核苷酸嵌套PCR(single oligonucleotide nested PCR,SON PCR)。本实验中介绍的是基于TaKaRa的试剂盒的实验方法。
基本原理是根据已知DNA序列分别设计3条同向且退火温度较高的特异性引物(SP Primer),以及退火温度较低的兼并引物,进行热不对称PCR反应。图3-8显示的是染色体步移技术的基本步骤。
【实验方法】
1.基因组DNA的获取。(方法参照实验十八)
2.已知序列的验证:根据已知序列设计特异性引物(扩增长度最好不少于500bp),对模板进行PCR扩增,然后对PCR产物进行测序,与参考序列比较,确认已知序列的正确性。
3.特异性引物的设计:根据验证的已知序列,按照前述的特异性引物设计原则设计3条特异性引物,即: SP1,SP2,SP3。
4.第一轮PCR反应:取适量基因组DNA作为模板,以AP Primer(4种中的任意一种,以下以AP1 Primer为例)作为上游引物,SP1 Primer为下游引物,进行第一轮PCR反应。
①按下列组分(表3-1)配制第一轮PCR反应液。
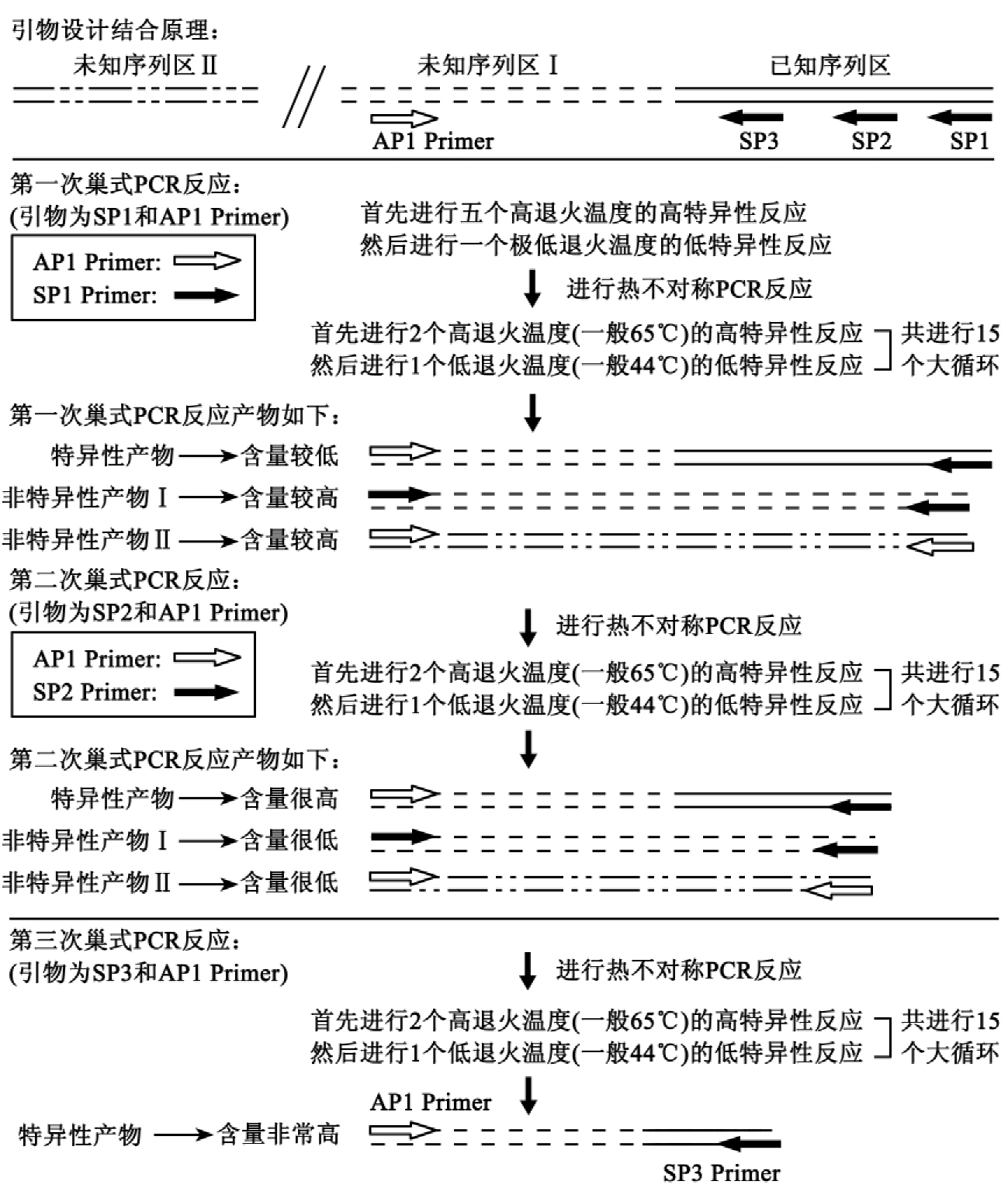
图3-8 染色体步移技术的基本步骤
表3-1 染色体步行中的第一轮PCR反应

②第一轮PCR反应条件:
94℃ 1分钟
98℃ 1分钟
94℃ 30秒
60~68℃ 1分钟 5循环
72℃ 2~4分钟
94℃ 30秒; 25℃ 3分钟; 72℃2~4分钟

72℃ 10分钟
5.第二轮PCR反应:将第一轮PCR反应液稀释1~1000倍后,取1μl作为第二轮PCR反应的模板,以AP1 Primer为上游引物,SP2 Primer为下游引物,进行第二轮PCR反应。
①按下列组分(表3-2)配制第二轮PCR反应液。
表3-2 染色体步行中的第二轮PCR反应

②第二轮PCR反应条件:

72℃ 10分钟
6.第三轮PCR反应:将第二轮PCR反应液稀释1~1000倍后,取1μl作为第三轮PCR反应的模板,以AP1 Primer为上游引物,SP3 Primer为下游引物,进行第三轮PCR反应。
①按下列组分配制第三轮PCR反应液(表3-3)。
表3-3 染色体步行中的第三轮PCR反应

续表

②第三轮PCR反应条件:

72℃ 10分钟
7.取第一轮、第二轮和第三轮PCR反应液各5μl,使用1%的琼脂糖凝胶进行电泳。
8.切胶回收清晰的电泳条带,以SP3 Primer为引物对PCR产物进行DNA测序。
【思考题】
简述染色体步行技术的基本原理。
免责声明:以上内容源自网络,版权归原作者所有,如有侵犯您的原创版权请告知,我们将尽快删除相关内容。













