实验十四 RNAi原理及基本步骤
【实验目的】
了解和掌握植物RNAi技术的原理及基本步骤。
【实验原理】
在研究植物发育过程中的基因功能时,常常通过降低目的基因在植株中的表达来探求基因功能。在植物体内降低基因表达的方法较为常用的是RNA干扰。
RNA干扰(RNA interference,RNAi)是指在进化过程中高度保守的、由双链RNA(double-stranded RNA,dsRNA)诱发的、同源mRNA高效特异性降解的现象。RNAi是在研究秀丽新小杆线虫(C.elegans)反义RNA(antisense RNA)的过程中发现的,由dsRNA介导的同源RNA降解过程。1995年,Guo等发现注射正义RNA(sense RNA)和反义RNA均能有效并特异性地抑制秀丽新小杆线虫par-1基因的表达,该结果不能使用反义RNA技术的理论做出合理解释。直到1998年,Fire等证实Guo等发现的正义RNA抑制同源基因表达的现象是由于体外转录制备的RNA中污染了微量dsRNA而引发,并将这一现象命名为RNAi。此后,dsRNA介导的RNAi现象陆续发现于真菌、果蝇、拟南芥、斑马鱼等多种真核生物中。植物中的转录后基因沉默(posttranional gene silencing,PTGS)、共抑制(cosuppression)及RNA介导的病毒抗性、真菌的抑制现象均属于RNAi。
RNAi有两种既有联系又有区别的途径,即siRNA(small interference RNA)途径和miRNA(microRNA)途径。siRNA途径是由dsRNA(double-stranded RNA)引发的,dsRNA被一种RNaseⅢ家族的内切核酸酶(Dicer)切割成21~26 nt长的siRNA,通过siRNA指导形成RISC蛋白复合物(RNA-induced silencing complex)降解与siRNA序列互补的mRNA而引发RNA沉默。而miRNA途径中的miRNA是本身存在于细胞内的、含量丰富的不编码小RNA,一般为21~24个核苷酸。miRNA由Dicer酶切割内源性表达的短发夹结构RNA(hairpin RNA,hpRNA)形成。miRNA同样可以与蛋白因子形成RISC蛋白复合物,可以结合并切割特异的mRNA而引发RNA沉默。
针对上面两种不同的RNAi途径,在植物通过转基因方法获得目的基因表达量下降的研究过程中,也有两种不同的方法。第一种方法是反义RNA技术。反义RNA技术是将目的基因的反义链构建在转基因载体上,然后转入植株中,反义链会与正义链互补结合形成双链RNA,双链RNA复合体易被RNA酶III降解成为35 nt左右的小RNA分子,然后通过序列互补与mRNA结合,从而导致mRNA降解,进而抑制基因在植物中的表达。这种方法由于抑制效率较低,目前较少使用。另外一种方法是针对miRNA设计的转基因载体。Waterhouse等(1998)和Wesley等(2001)研究发现,在强启动子的下游加上反向重复的cDNA片段,然后将该载体转入植物中,将会导致转基因植株中目的基因的表达下降。反向重复的cDNA片段转入植物基因组后,通过转录产生自我互补的hpRNA,Dicer酶切割hpRNA后形成miRNA,miRNA与蛋白因子形成RISC蛋白复合物,结合并切割特异的mRNA而引发RNA沉默。目前常用的植物RNAi载体是pKannibal质粒(图1-9,图1-10)。
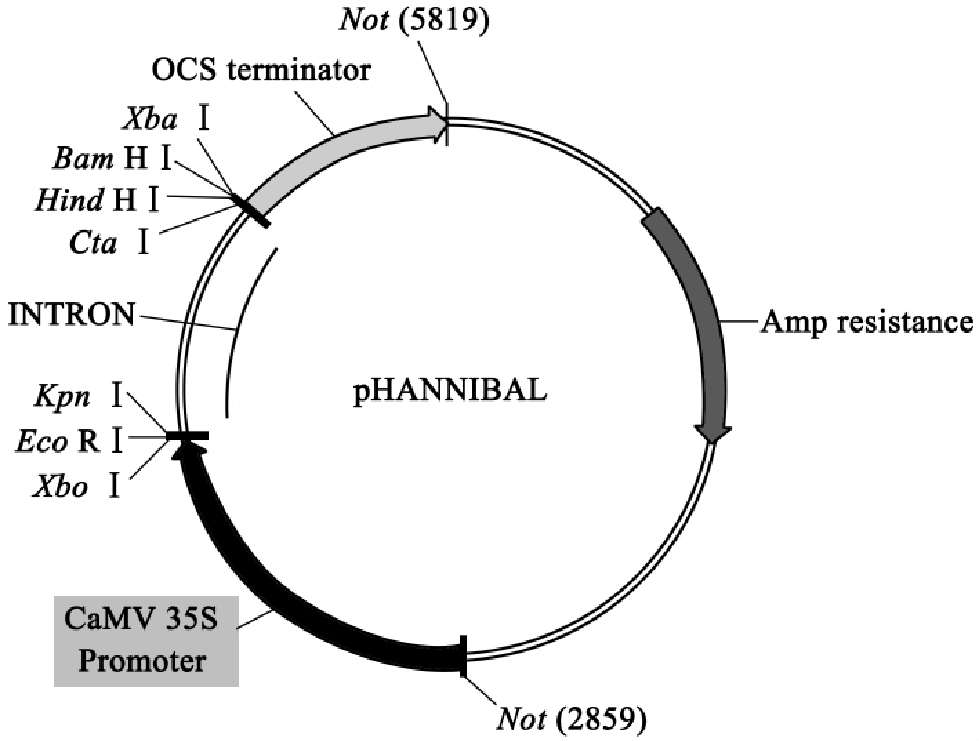
图1-9 pHANNIBAL图谱
【实验方法】
这里只列出步骤,具体的实验方案参照本书中的其他章节以及
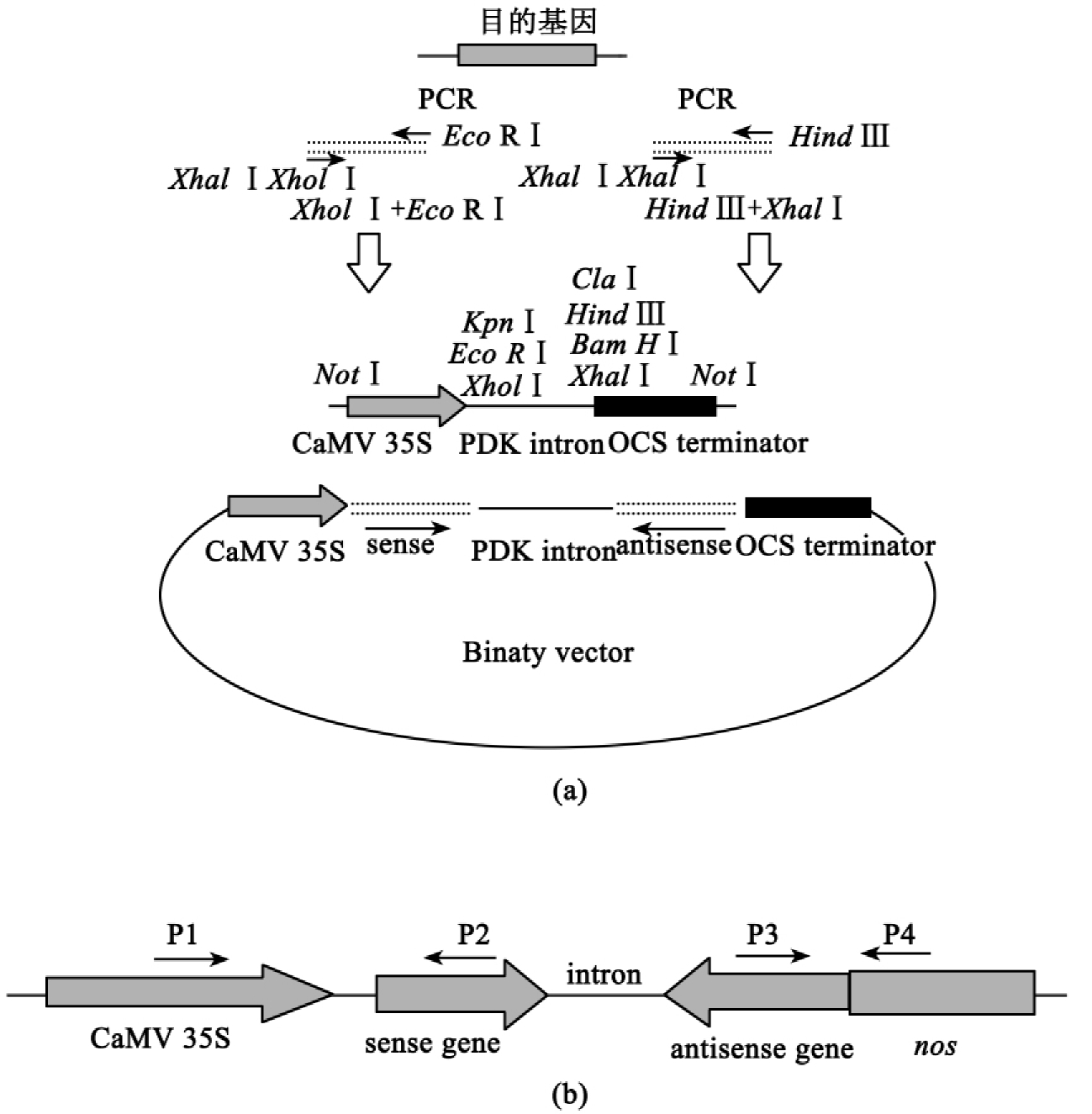
图1-10 pHANNIBAL构建(a)和验证(b)
分子生物学操作手册。
1.载体的选择。
2.从基因组序列中克隆目的基因,并测序验证。
3.选择合适的酶切位点,按照载体说明,将目的基因构建到载体中(图1-10(a))。验证基因是否构建到载体中的方法:一般利用载体上序列的特异引物P1和P4及基因本身特异引物P2和P3结合进行PCR验证,如图1-10(b)所示。
4.重组载体转化农杆菌。
免责声明:以上内容源自网络,版权归原作者所有,如有侵犯您的原创版权请告知,我们将尽快删除相关内容。














