实验使用的三种农药分别是:杜邦万灵(其有效成分为含量24%的灭多威可溶性溶剂)、氰戊菊酯(其有效成分为每100g制剂中含氰戊菊酯20g)、氧乐果(剂型为乳油,含量为40%),分别用蒸馏水配制成1∶50、1∶100、1∶500三种不同浓度的农药溶液。
实验样品来源于中国江西省赣州市宁都县某脐橙果园。选择没有表面缺陷、碰伤的脐橙共1 051个清洗并自然风干,然后采集脐橙最大横径处的光谱数据,把这些未喷施任何农药的脐橙数据作为对照组(数量为1 051个)。完成数据采集后把脐橙随机分成9组,分别喷施配制好的不同农药溶液。表6-5列出了10个组分别用于建模集和预测集的脐橙光谱数据个数,第1组为喷施杜邦万灵1∶50溶液的脐橙(数量为114个),第2组为杜邦万灵1∶100溶液的脐橙(数量为116个),第3组为杜邦万灵1∶500溶液的脐橙(数量为114个),第4组为喷施氰戊菊酯1∶50溶液的脐橙(数量为120个),第5组为喷施氰戊菊酯1∶100溶液的脐橙(数量为120个),第6组为喷施氰戊菊酯1∶500溶液的脐橙(数量为114个),第7组为喷施氧乐果1∶50溶液的脐橙(数量为117个),第8组为喷施氧乐果1∶100溶液的脐橙(数量为117个),第9组为喷施氧乐果1∶500溶液的脐橙(数量为117个)。在环境温度为10℃和相对湿度为60%的实验室条件下,把上述前三组脐橙放置168h后采集脐橙最大横径处的光谱数据。按每组脐橙总数约2∶1的比例随机抽取建模集与预测集样品,每组建模集与预测集样品数量如表6-5所示。因此,本实验中共分10个组,用于建模集和预测集的样品数目总数分别为1 400个和700个。
表6-5 每组脐橙样本数目及农药类型

遗传算法是模仿自然界生物进化机制发展起来的优化方法,作为一种实用、高效、鲁棒性强的优化技术,已为广大学者所重视。在近红外光谱检测应用中,遗传算法被用来对整个光谱区间进行优化,选择出最有效的特征区间或特征波长。
图6-9所示为建模集脐橙1 400个脐橙样本的原始光谱图,光谱范围为350~1 800nm。从图6-9中可以看出,在波段350~459nm区间的光谱曲线所包含的噪音较多,因此本实验中所选用的波段范围为460~1 800nm,共计1 341个波长数据。把整个区间分成40个子区间,除第40个子区间包含54个波长数据外,其余子区间均包含33个波长数据。用一个含有0.1且长度为40个字符的字符串S来表示40个子区间的选取,其中1和0分别表示其所对应的子区间是否被选取。在遗传算法中用适应度评估个体或解的优劣,适应度较高的个体遗传到下一代的概率较大,而适应度较低的个体遗传到下一代的概率相对小一些,评估个体适应度的函数称为适应度函数。
本实验应用支持向量机(SVM)对预测集数据进行识别的准确率为遗传算法的适应度函数,终止条件为达到最大的迭代次数。最佳的特征光谱区间是遗传迭代后识别准确率为最大值的区间组合。支持向量机的模型选择问题就是给定一个核函数,通过调节核参数和误差惩罚参数C来提高支持向量机训练精度,同时降低错误率,因此支持向量机的参数选择直接影响着其性能。
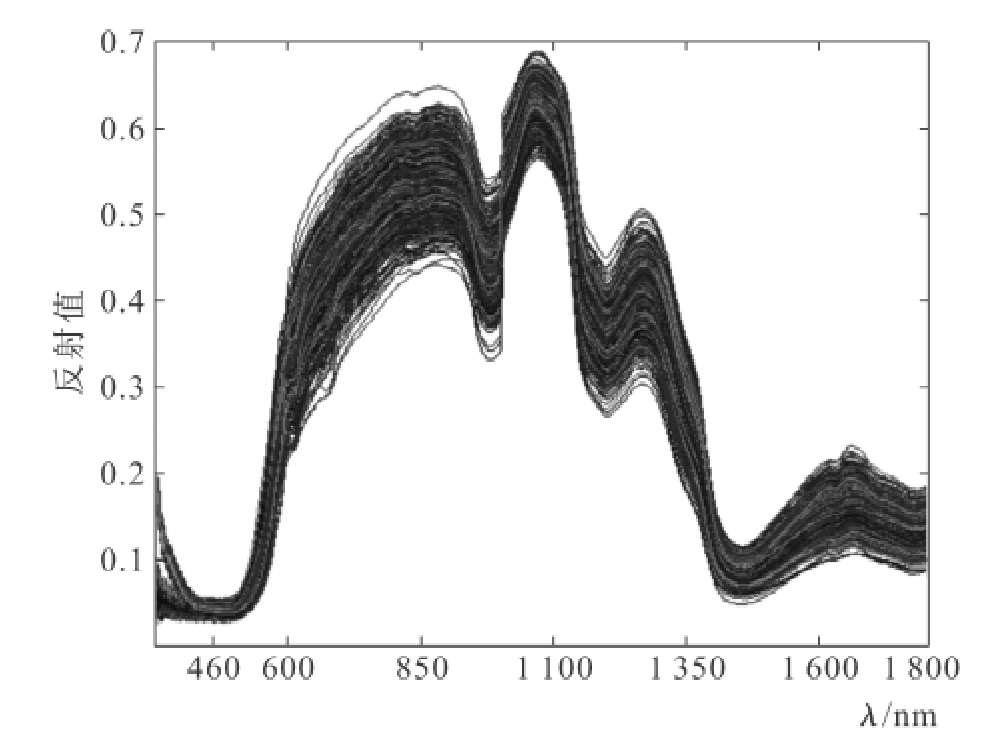
图6-9 建模集样本的原始光谱图
本实验建立模型所采用的核函数为径向基核函数。进行识别时,核函数参数γ和C的选择是一个重要问题,网格搜索法简单直接,因为每一个参数对(γ,C)都是独立的,可以并行地进行网格搜索,因此本实验采用网络搜索方法来选择最优的独立参数对(γ,C),以得到的最高准确率为评判依据,通过对全光谱的分析得到γ和C的取值分别为232与3。
本实验中GA的设定参量:区间数为40,初始群体为30,变量的二进制位数为40,代沟为0.9,交叉概率为0.7,遗传迭代次数为100。图6-10所示为经过100次迭代后的结果,实线表示每一代中最大的识别准确率,虚线表示每一代中所有个体识别准确率的平均值,可以看出在第99~100代识别准确率最大且值为98.86%。经过对这两代中最优个体的分析发现,其最佳特征区间均相同,其原因是在第99代中的最佳个体作为父辈被完整地遗传到了下一代中,因此最佳迭代数为第99代。
图6-11所示为第99代的最佳特征区间组合,图中灰色的条带区域表示此处的波段为被选中的5个特征区间,对应的波长数目为165个。

图6-10 每代中最大值识别准确率与识别准确率的均值的变化

图6-11 遗传算法结合支持向量机选取的最佳特征光谱区间
在被选中的5个特征区间的基础上,继续应用GA-SVM算法进行优化。此时GA的设定参量为:区间数为165,初始群体为40,变量的二进制位数为165,代沟为0.9,交叉概率为0.7,遗传迭代次数为100。图6-12所示为经过100次迭代后,每个变量(即特征波长)按照其被选取的次数所绘制的频率图。根据每个变量被选取的频率,由大到小重新排序,然后依次累加作为SVM的输入变量。图6-13所示为输入变量数与识别准确率变化曲线图,由图可以看出在变量数为85~95之间时,模型的识别准确率达到了最大值99.14%。因此,根据图6-13中识别准确率的变化趋势,最终确定输入变量数为85,而此时的识别准确率为99.14%。

图6-12 变量选取的频率图
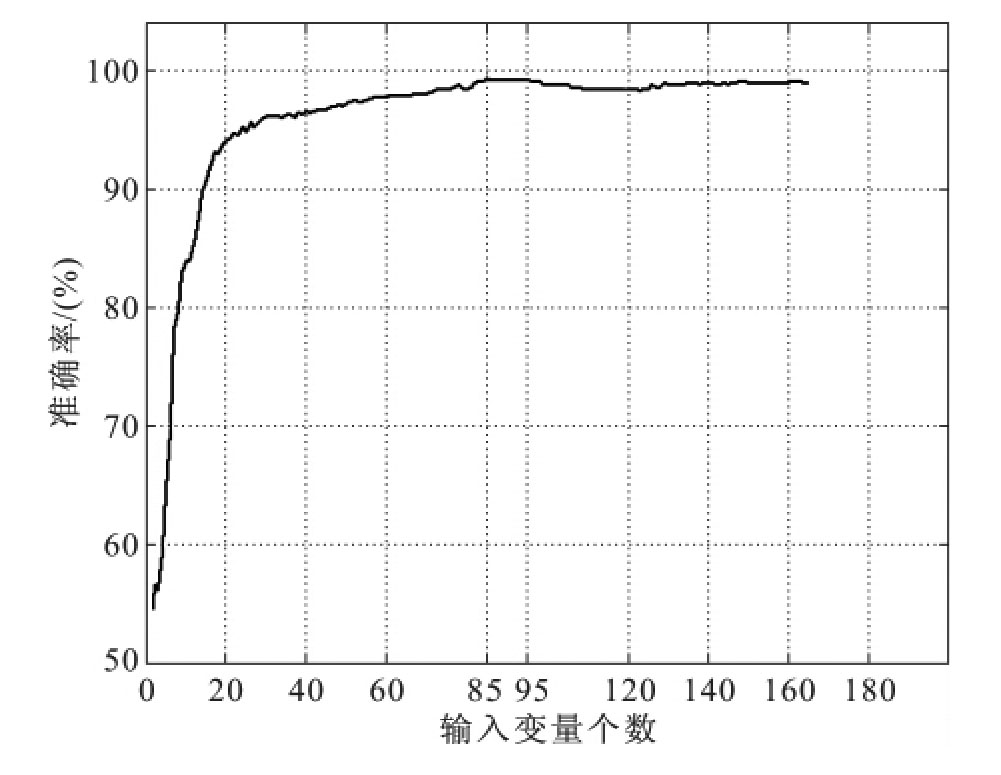
图6-13 识别准确率随变量选取的变化趋势
表6-6列出应用GA-SVM所建立的模型结果与仅应用SVM方法所建立的全光谱模型的比较结果。从表6-6中可以看出SVM的全光谱识别准确率较低,其值为83.43%,且SVM模型的输入变量数为1 341个,这使得模型的复杂度很高。使用GA-SVM所得到的模型建立在5个最佳特征光谱区间的基础之上(共165个光谱数据点),无论是模型的识别精度还是模型的简洁度都优于全光谱模型,而且对预测集中10个组别的识别结果的准确率为98.86%。因此,该方法不仅在降低模型复杂度(模型的输入参数为165个)上效果明显,而且其模型的识别精度也得到大幅度的提高。在选定的5个最佳特征光谱基础上,应用GA-SVM方法对模型的输入变量进行进一步的筛选,得到特征波长为85个,并且模型的识别能力还略有提高,说明应用GA-SVM方法所建立的识别模型比全光谱模型更加稳定、简洁。
表6-6 不同选择方法下应用SVM方法的检测结果
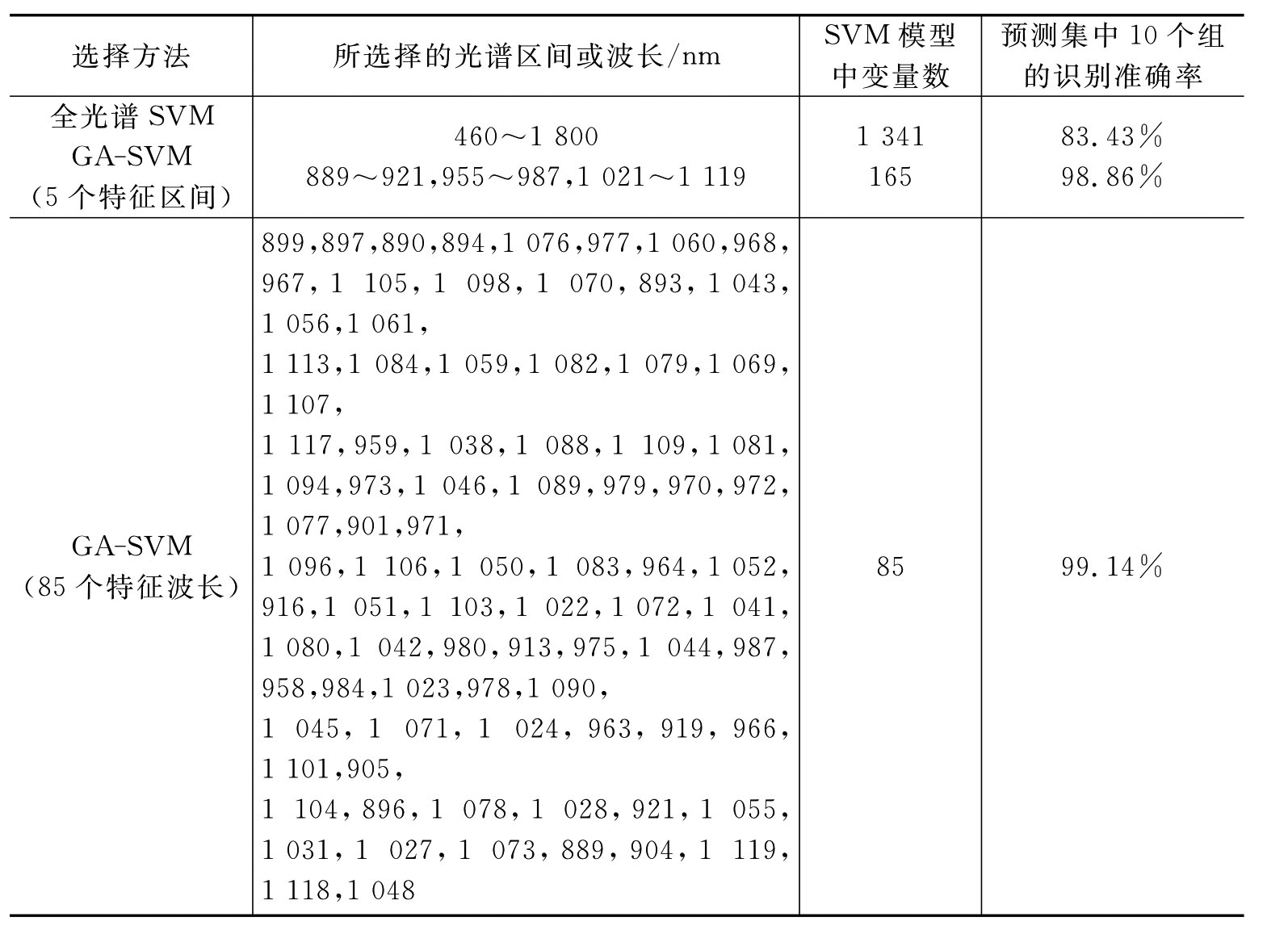
图6-14显示了波段范围为430~1 000nm,经一阶微分预处理后预测组中四种不同浓度农药污染样品真实值与预测值的检测结果图。图6-14中横坐标的1、2、3和4分别代表高浓度农药污染的脐橙样品、中浓度农药污染的脐橙样品、低浓度农药污染的脐橙样品和未被农药污染的脐橙样品,纵坐标为预测结果。

图6-14 被不同浓度农药污染样品真实值和预测值结果
免责声明:以上内容源自网络,版权归原作者所有,如有侵犯您的原创版权请告知,我们将尽快删除相关内容。

















