实验二十三 应用基因芯片研究酵母菌热激反应基因表达谱的变化
Ⅰ 样品标记
一、实验目的
用Cy3和Cy5荧光素分别标记待检测酵母菌的RNA反转录样品,用于芯片的杂交实验。
二、实验原理与方法
酵母属于真核生物,因此,在检测基因表达谱变化时,可以用Oligo(dT)15为引物和mRNA的poly(A)+互补,通过反转录合成cDNA第一条链,并在cDNA的合成过程中分别掺入Cy3-dCTP和Cy5-CTP荧光素。
三、酵母热激
酿酒酵母(Saccharomyces cerevisiae)菌株S288C在25℃条件下,YPD液体培养至OD600=0.60(OD600可在0.4~0.8之间,一般不要超过1.5,否则细胞的基因表达谱会受到营养条件的限制而发生改变),将培养物分成两等份,一份继续在25℃培养,另一份在40℃下热激处理0.5h,然后提取两种培养细胞的总RNA。
四、酵母总RNA抽提(以下操作请戴上手套)
RNA提取采用热酸酚法,具体操作如下。
1.实验材料和试剂:
(1)TES溶液(10mmol/L的Tris-HCl,pH7.5,10mmol/L的EDTA,0.5%SDS)的配制:在80mL水中加入1mL1mol/L的Tris-HCl(pH7.5),2mL0.5mol/L的EDTA,5mL10%的SDS,定容至100mL,高压灭菌。
(2)水饱和酚:4℃避光保存。
(3)NaAc:用DEPC水配制3mol/L NaAc,用醋酸调pH5.2,44.482N(10磅)高压灭菌。
(4)DEPC:Sigma公司产品。
(5)氯仿、无水乙醇:分装100mL棕色试剂瓶中写好标签,为RNA提取专用,4℃下保存。
(6)Nucleo-Spin RNA试剂盒(德国MN公司产品)。
RNA试剂盒(德国MN公司产品)。
2.实验步骤:
(1)将对照和经热激处理的酵母菌培养液分装在1.5mL的EP管中,室温下12000rpm离心2min,弃上清液,然后各加入1mL无菌水重悬,室温12000r/min离心2min,弃上清液。
(2)向沉淀菌体加入400μL的TES液,混匀后加入400μL的水饱和酚,充分振荡2min,混匀,在65℃的水浴中加热1h,期间每隔约10min振荡一次。
(3)4℃冰水浴5min后,12000rpm离心8min。
(4)将上层水相转移至新的1.5mL EP管中,加入400μL氯仿,充分振荡混匀,4℃条件下12000rpm离心8min。
(5)上层水相再转移至一新的1.5mL EP管中(约400μL),加入1/10体积的DEPC水配制的NaAc(3mol/L,pH5.2)和2.5倍体积的无水乙醇,在-20℃的冰箱中放置30min,4℃下12000rpm离心15min。
(6)弃上清液,向沉淀加入1mL的75%乙醇,4℃下12000rpm离心5min。
(7)弃上清液,将沉淀在室温晾干,最后加入50~100μL DEPC水,充分溶解RNA沉淀。
(8)取1μL上述RNA溶液,稀释100~200倍,测定260nm和280nm紫外光的吸收值。一般10mL OD600=0.60的培养液可提取400~700μg的总RNA。
(9)甲醛变性凝胶电泳检测RNA是否发生降解,见图23-1。

图23-1 RNA甲醛变性凝胶电泳图谱
(10)经电泳检查后确认RNA样品合格,然后用德国MN公司生产的Nucleo-SpinR RNA试剂盒纯化RNA(RNA纯化方法详见附录4.1)。对纯化后的RNA进行定量。
RNA试剂盒纯化RNA(RNA纯化方法详见附录4.1)。对纯化后的RNA进行定量。
五、RNA反转录直接荧光标记
1.实验材料和试剂:
(1)3-dCTP和Cy5-CTP(美国GE Health产品):各25nmol/L,-20℃避光保存。
(2)100mmol/L dNTP(美国Promega公司产品):将其配制成本实验用工作液:dATP∶dGTP∶dTTP∶dCTP为10mmol/L∶10mmol/L∶10mmol/L∶1mmol/L,-20℃保存。
(3)SuperscriptⅡ反转录酶(美国Invitrogen公司产品):200U/μL。
(4)5×1st strand buffer(美国Invitrogen公司产品):0.1mol/L DTT。
(5)Oligo(dT)15(美国Promega公司产品):-20℃保存,用DEPC-H2O溶解为1μg/μL。
(6)外标原液(Y1~Y8)(北京博奥生物有限公司产品)。
(7)反转录终止液(35mmol/L pH8.0的EDTA、1.4mol/L NaOH)配制1.4mL;取0.5mol/L EDTA(pH8.0)100μL,10mol/L NaOH 200μL,加灭菌去离子水1100μL至1.5mL离心管中,4℃保存。
(8)NucleoSpin Extract Ⅱ Kit(德国MN公司产品)。
Ⅱ Kit(德国MN公司产品)。
(9)10mol/L乙酸:取冰醋酸(浓度为17.4mol/L)0.574mL,加灭菌去离子H2O0.426mL于1.5mL离心管中,4℃保存。
2.实验方法(以下操作请戴上手套):标记探针的具体步骤如下。
(1)在两个0.2mL EP离心管中分别加入以下试剂,配制对照组(X体系)和热激实验组(Y体系)试剂,总体积均为20μL:
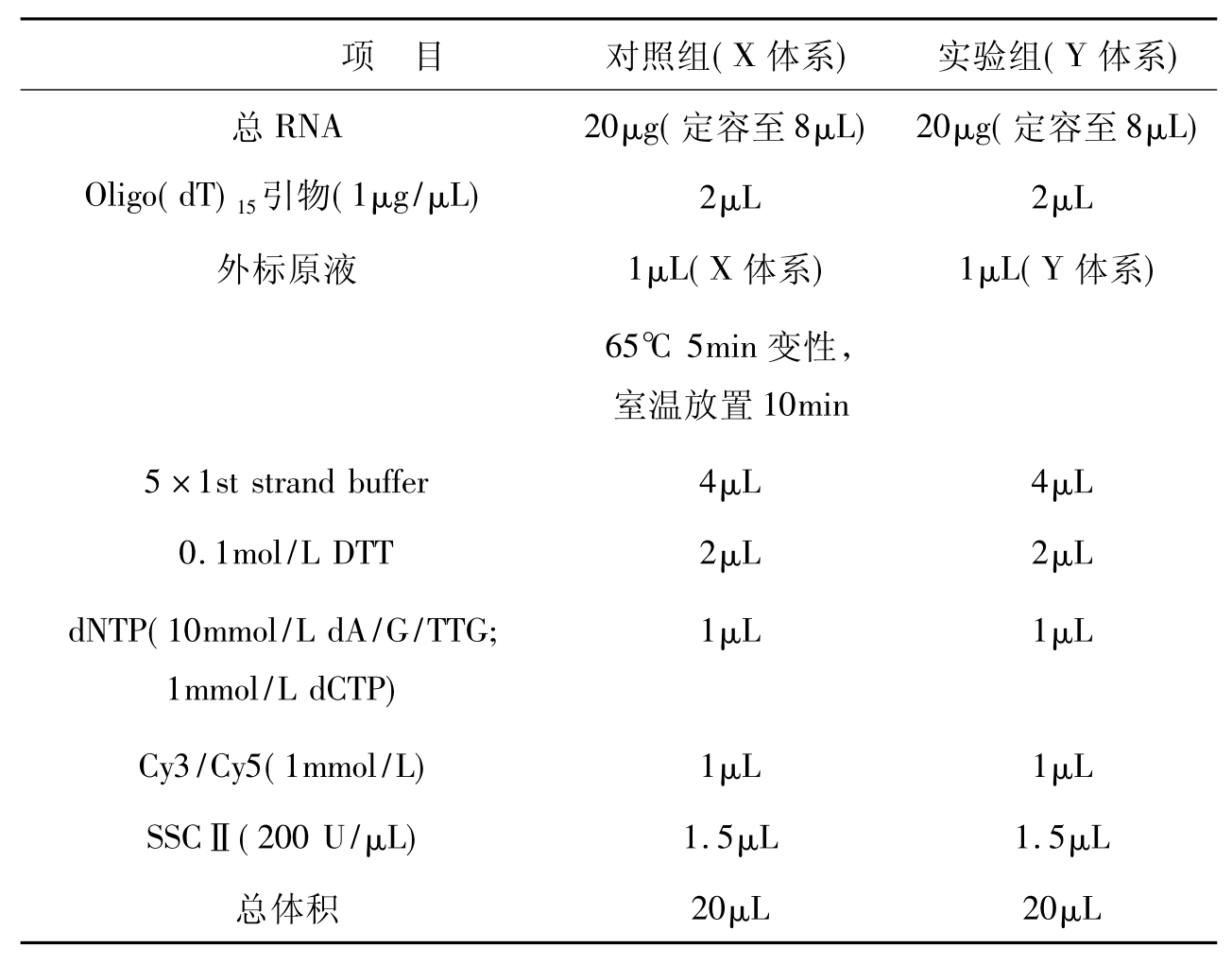
(2)将上述反应液用枪头小心吹打、混匀,用掌上离心机瞬时离心,在42℃空气浴反应1.5~2h后,冰浴2min。
(3)加入5μL终止液,在70℃下反应10min(消化反应液中的RNA),加入1μL10mol/L乙酸。
(4)上述合成的荧光标记的cDNA(探针)用NucleoSpin Extract PCR纯化柱纯(cDNA纯化方法详见本实验附录2),Elution缓冲液洗脱,洗脱液使用量约为60μL。
PCR纯化柱纯(cDNA纯化方法详见本实验附录2),Elution缓冲液洗脱,洗脱液使用量约为60μL。
(5)测定OD260、OD280、OD550或OD650。Cy3和Cy5分别在OD550和OD650有最大光吸收。将纯化后的探针(约60μL)全部转移到100μL比色杯中,测定OD550或OD650的光吸收。
(6)计算Cy3、Cy5的掺入量:
Cy3的总掺入量(pmol)=OD550×总探针体积(μL)÷0.15
Cy5的总掺入量(pmol)=OD650×总探针体积(μL)÷0.25
(7)将纯化过并已定量的标记样品,放在真空旋转浓缩仪中抽干(在抽干过程中要避光),加入杂交液溶解后用于杂交。
Ⅱ 杂交反应
一、实验目的
将样品与探针杂交,使其与能够互补配对的DNA链结合。
二、实验原理与方法
基本原理参照实验二十二。
三、实验材料和试剂
1.十二烷基硫酸钠(SDS)(美国Sigma公司生产)。
10%SDS的配制:在900mL水中溶解100g电泳级SDS,加热至68℃助溶,加入几滴浓盐酸调节溶液的pH值至7.2,加水定容至1L,分装备用。
2.柠檬酸三钠、氯化钠、醋酸钠、无水乙醇(均为国产分析纯试剂)。
3.甲酰胺(formamide)(美国Sigma公司生产)。
4.20×SSC的配制:在800mL水中溶解175.3g NaCl和88.2g柠檬酸钠,加入数滴HCl溶液调节pH值至7.0,加水定容至1L,分装后高压灭菌。
5.50×Denhart溶液。
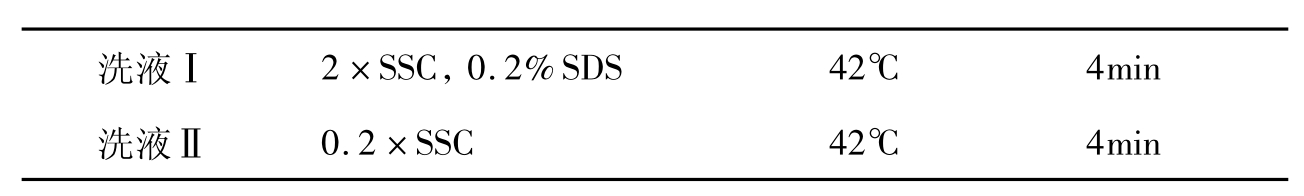
6.洗液Ⅰ:2×SSC,0.2%SDS。
洗液Ⅱ:0.2SSC。
四、实验步骤与结果
1.基因芯片的预处理(以下操作请戴上手套):
(1)挑选扫描预选合格的芯片,将点有DNA的一面向下,芯片距水面距离为2~3cm,在60℃水浴锅上水合10s,在空气中室温干燥,再进行一次水合。
(2)将点有DNA样品的一面朝上,放在紫外交联仪中250mJ交联。
(3)将芯片放在42℃预热,0.5%SDS清洗10min,水平摇床80rpm晃动。
(4)将芯片放在42℃预热,蒸馏水中清洗4~5min,水平摇床80rpm晃动。
(5)将芯片放在无水乙醇中清洗1~2min,水平摇床80rpm晃动。
(6)把芯片放在50mL锥形离心管中,1500rpm离心1min,以除去芯片表面的液体,此时的芯片即可用于杂交。
2.芯片杂交:
配制杂交液(12μL体系)
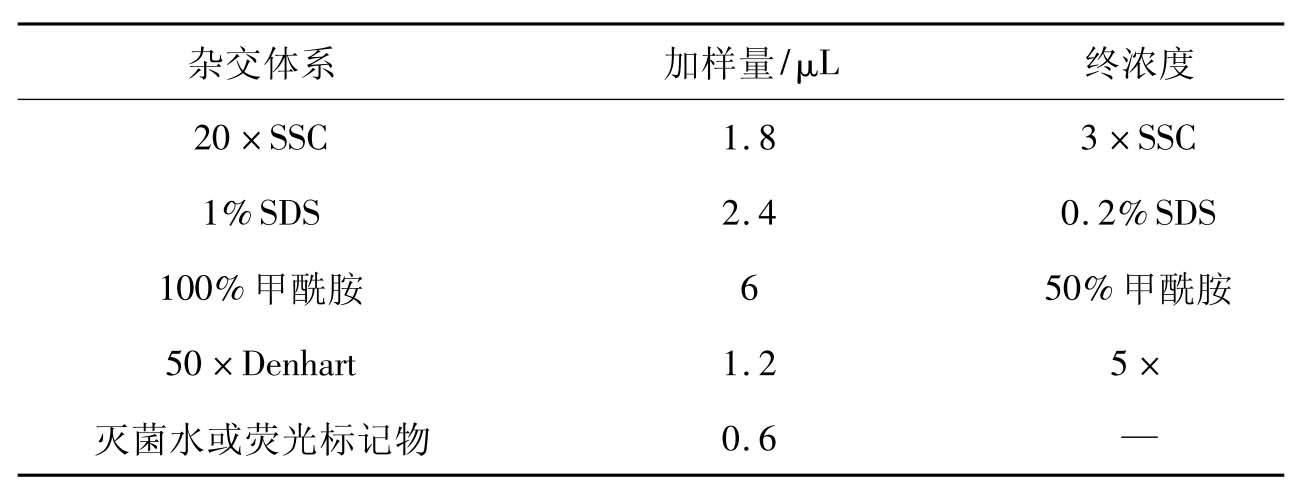
杂交面积7mm×7mm,杂交体积为12μL,直接标记需要的荧光掺入量大于12pmol(配制含荧光标记物的杂交液时,可先取6μL甲酰胺溶液放入探针标记物管中,溶解样品,短暂离心,再将溶解液转移至另一探针标记物中溶解,混匀后短暂离心,将溶液转移到0.2mL的EP管中,再加入其他杂交试剂,短暂离心)。95℃热变性3min,冰浴骤冷,如果有SDS析出,待其在室温中溶解后,再用于杂交。
芯片杂交的操作过程如下:
(1)贴围栏:撕开围栏中的不干胶面和贴纸,用镊子夹住围栏放在特制的杂交围栏模具中,有粘性的一面朝上。将芯片无标签的一端顶在模具的内侧,手持芯片有标签一端缓缓放下,使芯片与围栏贴紧。注意:芯片上有样品的一面向下。(详见实验二十二)
(2)打开杂交盒,向杂交盒内的凹槽内加入200μL蒸馏水,然后把贴好围栏的芯片放入杂交盒内,点有样品的一面朝上。将带有凸块的盖玻片卡放在芯片的黑色围栏上,注意有凸块的一面对着芯片;然后从盖玻片的小孔缓缓加入12μL的杂交液,杂交液会凭借液体表面张力在盖玻片下面的凸面和芯片之间形成一道液膜。盖上杂交盒盖,分别将两个金属夹卡进两侧,动作要平缓,不要震动盖玻片或芯片以避免破坏液膜。快速把杂交盒放入42℃水浴。(详见实验二十二)杂交12~14h(过夜)。
3.芯片清洗(详见实验二十二):
(1)将洗液Ⅰ、洗液Ⅱ放在42℃水浴锅中预热。
(2)杂交结束后,将芯片转移到盛放洗液的清洗盒中(围栏可保留),杂交面朝上,放在水平摇床上缓慢清洗。清洗条件如下:
(3)芯片清洗后,放在50mL锥形离心管中,1500rpm离心1min,除去玻片表面的液体,下一步进行芯片扫描。
Ⅲ 共聚焦扫描及结果分析
一、实验目的
将生物芯片杂交信息的图像阅读出来,转变为可供计算机处理的数据;对扫描图像利用计算机软件进行图像处理、数据提取、统计分析和生物学分析。
二、实验原理
激光共焦芯片扫描仪以激光作为激发光源,荧光物质被激发后可以产生较高强度的发射荧光,可大大提高检测灵敏度。由于cDNA芯片技术常用来检测基因的差异性表达,靶核酸分子常常用两种荧光染料标记,故目前商业化的DNA芯片扫描仪也多采用两种不同波长的激光器作为发光源,以激发不同荧光染料标记的靶分子。DNA芯片扫描仪通常采用激光共焦显微镜原理进行设计。
由激光器发射出来的激光经由A、B透镜组成的扩束系统被扩展成一定口径的平行光束,经激光窄带干涉滤光器C(带宽通常为几十纳米),过滤除去其他波长的光,以降低检测背景。激光由双色镜D反射至物镜组E,双色镜的作用是让入射激光被反射,而激发出来的荧光可以通过而不会被反射。物镜组E将激光聚焦在DNA芯片上,聚焦后的光斑一般在几微米到几十微米之间。标记有荧光染料的靶分子在激光激发下所产生的荧光由物镜组E捕获后变成平行光,通过双色镜D,由反射镜F反射至窄带干涉滤光器G,以滤除除发射荧光以外的光,再由透镜组H将其聚焦在共焦光阑I上,通过光阑的光最后由光电倍增管PMT检测,经放大器滤波放大,放大信号由模数转换器将模拟信号转换成数字信号后,由计算机采集存储。共焦光阑的光孔设计得较小,目的是使正确聚焦在芯片表面上的激光所诱导的荧光能由透镜组H聚焦在光阑中,通过光孔被检测出。而芯片表面下部基质或芯片表面上方尘埃所产生的荧光或杂散光而不能聚焦在光阑上,大部分不相关的光被光阑阻挡不能通过,可大大减少由于基质和灰尘产生的背景荧光。扫描由电动机驱动的芯片载台作X-Y二维移动来实现。扫描的结果由计算机裁决并以TIFF或BIP等图像文件格式变为16位文件存储。扫描得到的图像是黑白的,包括了图像的灰度信息。图像的灰度信息可由计算机软件处理后代表不同的杂交信号强度。多种荧光染料标记的芯片可以通过切换不同波长的激光和不同波长范围的干涉滤光器C和G,重复扫描来分别测定。
三、实验器材与试剂
晶芯LuxScan10K-A双通道激光共聚焦扫描:北京博奥生物有限公司产品。
SpotData软件:北京博奥生物有限公司产品。
四、实验步骤
共聚焦扫描:按如下方式操作LuxScan10K-A双通道激光共聚焦扫描仪。
(1)启动计算机工作站。
(2)用USB连接线将扫描仪和计算机连接起来,打开扫描仪电源。Windows桌面任务栏将出现USB设备图标。
(3)启动LuxScan10K-A应用程序。程序开始搜索扫描仪和系统自检。
(4)系统自检无误,应用程序进入主界面。
(5)打开红色、绿色两个激光器进行预热(10min)。
(6)将芯片平稳放置在托架小片,水平轻缓地推入扫描仪窗口,载入芯片。
(7)设定扫描区域和扫描参数。
(8)双通道扫描,在扫描第二个通道时,会同时产生合成图像,是两个通道图像叠加后的结果。
(9)逐个保存扫描图像,当需要打开这些图像时,用鼠标点击拖拉,或按住Ctrl键时同时用鼠标点击可以选中多个要打开的图像。同时打开同一区域的两个通道,扫描图像文件会自动合成图像,合成图是伪彩渲染的图像,仅供直观显示,不能保存为TIFF格式图像文件供进一步数据分析。
在本实验中,我们使用Cy5来标记40℃处理的样品,使用Cy3来标记25℃的参照样(或者进行荧光交换)。在进行前面的第8步扫描操作时要以外标Y1~Y4所在位置进行Cy3和Cy5荧光通道的平衡,选择合适的激光功率(laser power)和光电倍增系数(PMT Gain)设置使其Cy5和Cy3信号的比值约为1.0,此过程也叫硬件归一化。平衡后的扫描条件是,Cy5和Cy3激光强度分别为82和75,Cy5通道的光电倍增系数(PMT Gain)为80%,Cy3通道的光电倍增系数为70%。
五、结果分析
扫描图像利用博奥生物有限公司的SpotData软件进行数据提取,对外标Y1~Y4的强度值做线性归一化,计算归一化之后的荧光强度比值(本实验中就是Cy5/Cy3的比值)。
如果荧光强度比值的数值大于2,则表示该基因在热激条件下的表达发生了上调;强度比值数值小于0.5,则表示该基因在热激条件下的表达被抑制。表23-1就是这些差异表达基因的荧光强度比值。
表23-1 差异表达基因列表
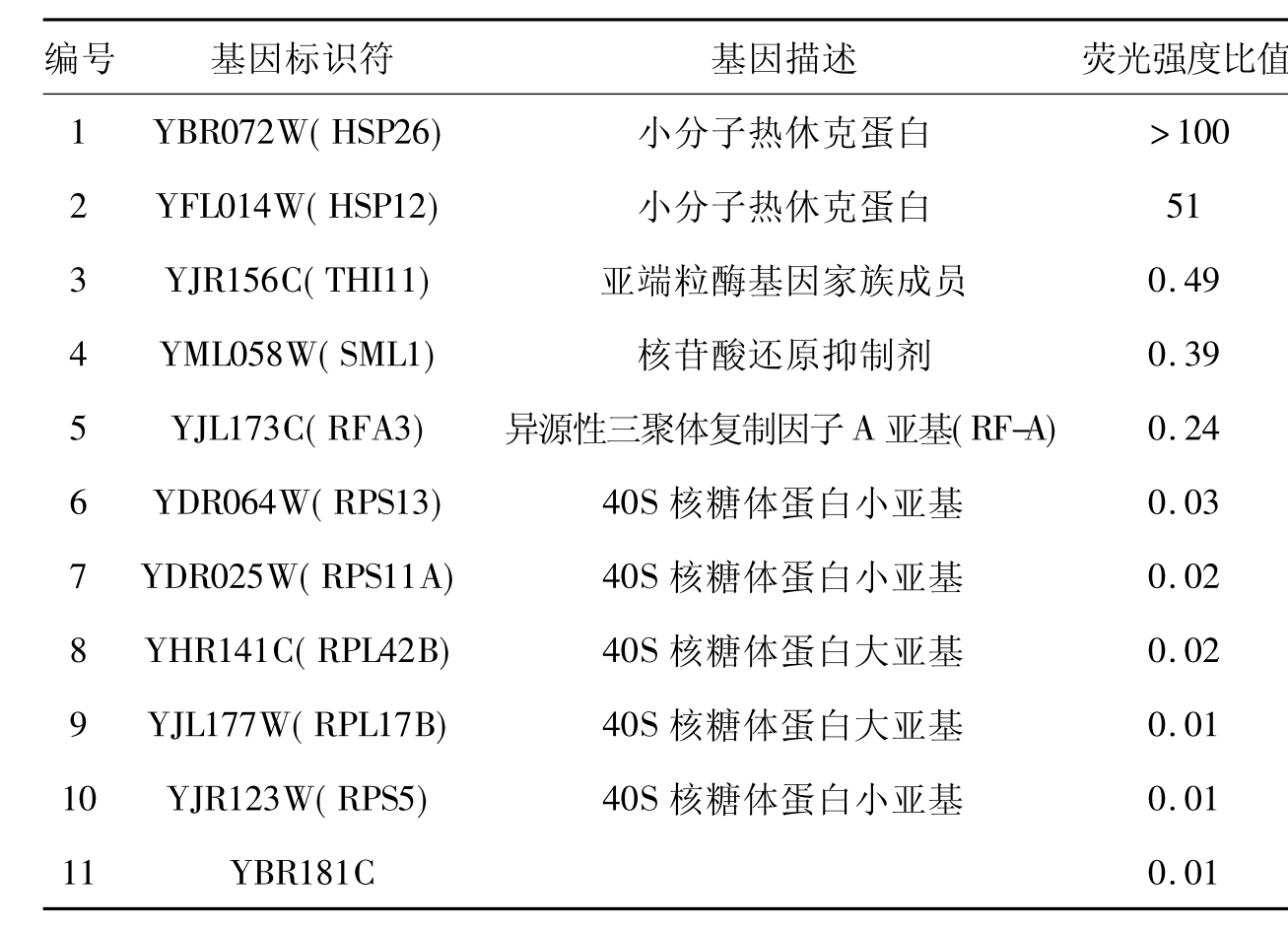
从表23-1中可以看出,酵母细胞受到热刺激后,HSP26和HSP12的基因表达量分别被强烈诱导了。此外,多个核糖体蛋白的基因表达量下调,表明细胞通过减少蛋白质的合成,把节约的能量用到了细胞的保护性反应中,以克服恶劣环境所带来的不良影响。
免责声明:以上内容源自网络,版权归原作者所有,如有侵犯您的原创版权请告知,我们将尽快删除相关内容。















